Wysokorozdzielcze struktury ludzkiego Elongatora – układu białek odpowiedzialnego za ich produkcję - uzyskali naukowcy z Uniwersytetu Jagiellońskiego. Okazało się, że cząsteczki tRNA ze zmodyfikowaną chwiejną urydyną wiążą się z rybosomami zapewniając poprawne fałdowanie nowo powstających białek.
Elongator jest kluczowy do tego, aby produkcja białek w organizmach wielokomórkowych zachodziła w poprawny sposób. Skomplikowany układ białek składa się z wielu podjednostek, a mutacje wpływające na kształt lub aktywność którejś z nich powodują jego dysfunkcje.
Na poziomie całego organizmu takie mutacje objawiają się u ludzi w postaci różnych chorób i niepełnosprawności, w tym intelektualnej. Więcej na ten temat tu.
Elongator odpowiada za wprowadzanie pewnej bardzo małej grupy chemicznej w konkretne miejsce w antykodonie tRNA. Obecność tej chemicznej modyfikacji jest istotna w procesie produkcji białek. Elongator modyfikuje tzw. chwiejną urydynę antykodonu. Mutacje genomowe w Elongatorze wywołują poważne choroby u ludzi.
Zrozumienie mechanizmu działania Elongatora było celem Grupy Badawczej Maxa Plancka z Małopolskiego Centrum Biotechnologii (MCB) UJ w Krakowie. Badania zespołu dr hab. Sebastiana Glatta zostały przeprowadzone we współpracy z naukowcami z Uniwersytetu w Kassel i Uniwersytetu Technicznego w Berlinie. Do obserwacji Elongatora naukowcy zastosowali krio-mikroskop elektronowy Titan Krios G3i, znajdujący się w Narodowym Centrum Promieniowania Synchrotronowego SOLARIS.
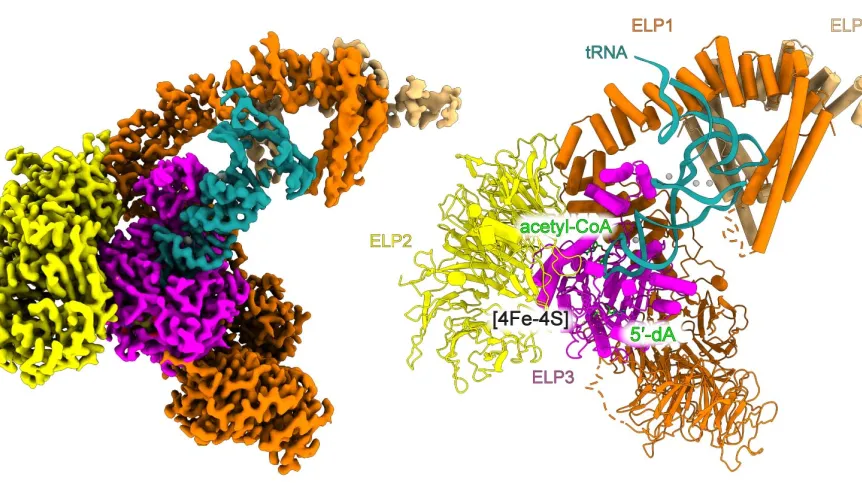
Jak się okazało, Elongator składa się z dwóch odrębnych podkompleksów: Elp123 i Elp456. Grupa karboksymetylowa wprowadzona przez Elongator służy jako podstawa do syntezy kolejnych powiązanych modyfikacji. Cząsteczki tRNA ze zmodyfikowaną chwiejną urydyną wiążą się optymalnie z rybosomami zapewniając poprawne fałdowanie nowopowstających białek.
Zespołowi MCB udało się określić strukturę ludzkiego Elongatora o najwyższej dotąd opublikowanej rozdzielczości (2,9 Å). Przedstawia ona podkompleks Elp123 w kompleksie z tRNA i cząsteczką acetylo-CoA oraz organizację miejsca aktywnego wraz z pętlą antykodonu tRNA, która zawiera cel modyfikacji, mianowicie chwiejną urydynę. Struktura ta pozwoliła zidentyfikować nieprzewidzianą rolę innej powszechnie zachowanej dla aktywności Elp123 urydyny, która znajduje się obok chwiejnej urydyny. Badanie wraz z opisem mechanizmu działania elongatora zostało opublikowane w Nature Communications. Praca przedstawia także dodatkowe struktury obrazujące kompleks podczas różnych pośrednich etapów reakcji modyfikacji.
Autorzy zidentyfikowali również szereg zachowanych reszt aminokwasowych zlokalizowanych w miejscu aktywnym podjednostki katalitycznej, które są niezbędne do przeprowadzenia reakcji chemicznej. Wszystkie odkrycia zostały zweryfikowane i poparte eksperymentami in vivo i in vitro.
„Naszym celem zawsze było zrozumienie, jak ten klinicznie ważny kompleks białkowy działa u ludzi. Nasza wysoko-rozdzielcza struktura ludzkiego Elp123 pozwala precyzyjnie i bezpośrednio wskazać położenie mutacji, które prowadzą do zaburzeń zdrowotnych związanych z Elongatorem i spróbować zrozumieć ich wpływ na aktywność kompleksu” – podsumowuje dr hab. Sebastian Glatt, współautor artykułu.
Badania przeprowadzono z funduszy Programu Horyzont 2020 ERC i Fundacji na rzecz Nauki Polskiej.
Nauka w Polsce
kol/ agt/
Fundacja PAP zezwala na bezpłatny przedruk artykułów z Serwisu Nauka w Polsce pod warunkiem mailowego poinformowania nas raz w miesiącu o fakcie korzystania z serwisu oraz podania źródła artykułu. W portalach i serwisach internetowych prosimy o zamieszczenie podlinkowanego adresu: Źródło: naukawpolsce.pl, a w czasopismach adnotacji: Źródło: Serwis Nauka w Polsce - naukawpolsce.pl. Powyższe zezwolenie nie dotyczy: informacji z kategorii "Świat" oraz wszelkich fotografii i materiałów wideo.













